基于Web技术的蛋白质复合物识别系统的设计与实现毕业论文
2020-03-30 12:18:40
摘 要
随着Web技术的快速发展和互联网带来的便捷,越来越多的用户趋向于用Web技术来解决问题,为了进一步满足用户的需求,蛋白质复合物识别系统的发展也势在必行。根据当前ppi网络与用户对参数文件的要求,设计与实现一个具有基本蛋白质复合物识别功能的在线蛋白质复合物识别系统很有必要。
本次设计,是基于jsp servlet模式的蛋白质复合物识别系统的设计与实现,目的是利用互联网带来的便捷性,基于已发表的蛋白质复合物识别方法来设计和实现在线蛋白质复合物识别系统,并通过互联网向科研人员开放。通过前期对系统的业务功能需求及非功能性需求的分析,确定此次系统的基本功能后,着手建立了一个更新数据库、配置参数文件、调用程序运行、向用户发送邮件、将结果可视化等功能的蛋白质复合物识别系统。
该系统通过采用D3.js对数据可视化,使得数字的图形化展现地更加简单鲜活。
关键词:蛋白质复合物识别系统;jsp servlet模式;D3.js数据可视化
Abstract
With the rapid development of Web technology and the convenience brought by the Internet, more and more users tend to use Web technology to solve problems, In order to further meet the needs of users, the development of protein complex recognition system is also imperative.
It is necessary to design and implement an online protein complex identification system with the basic protein complex recognition function according to the requirements of the current PPI network and the user's parameter files.
This design is the design and implementation of protein complex recognition system based on jsp and servlet mode. The purpose is to design and implement an online protein complex identification system based on the published protein complex identification method and open to researchers through the Internet, using the convenience of the Internet. After the analysis of the functional requirements and non functional requirements of the system, the basic functions of the system are determined, and a protein complex identification system is set up to update the database, configure the parameter files, call the program, send the mail to the user, and visualize the results.
The system makes use of D3.js to visualize data, making digital graphics more simple and vivid.
Key Words: protein complex identification system;jsp and servlet mode;D3.js data visualization
目 录
摘 要 I
Abstract II
第1章 绪论 1
1.1 研究背景 1
1.2 研究目的及意义 1
1.3 国内外研究现状 1
1.4 课题研究内容 2
第2章 DCAFP算法介绍 3
2.1 算法背景 3
2.2 算法思想及实现 5
第3章 系统设计 7
3.1 系统描述 7
3.2 系统主要功能及实现 8
3.2.1更新数据库功能 8
3.2.2配置参数文件功能 9
3.2.3调用程序运行功能 10
3.2.4给用户发送邮件功能 10
3.2.5可视化功能 10
第4章 系统测试及结果 13
4.1 文件上传界面 13
4.2 更新数据库功能 14
4.3 配置参数文件功能 15
4.4 调用程序运行功能 16
4.5 给用户发送邮件功能 17
第5章 结论 22
第1章 绪论
1.1 研究背景
预测蛋白质相互作用网络中的复合体和功能模块对于理解生物系统的组织和功能具有重要的意义[[1]]。很多计算方法因此被应用于识别蛋白质-蛋白质相互作用网络中的蛋白质复合物。除了可以被计算方法采用用于识别蛋白质复合物的信息外,还有PPI网络中的图拓扑结构(因为蛋白质的拓扑结构能够告诉我们蛋白质的功能和组织信息[[2]]),把蛋白质的功能信息考虑在内现在变得很流行。相关的方法实现依赖于这样的思想:同一个蛋白质复合物中的蛋白质和相似的功能信息有关系。然而,我们在以前的研究中发现对于大多数蛋白质复合物来说他们的蛋白质只是在一些功能种类的子集类似而不是所有功能,因此如果识别蛋白质复合物的时候把每个功能种类的参数考虑在内,结果准确性将会提升,本系统中所使用的DCAFP算法正是这样一种算法,它同时考虑了蛋白质的功能信息和ppi网络的拓扑结构,使得结果更为精准。
1.2 研究目的及意义
(1)综合了已发表的各种蛋白质复合物识别方法的优缺点,寻找出一种使得结果更为精准和更有意义的方法。
(2)设计成在线蛋白质复合物识别系统,并通过互联网向科研人员开放,相比于传统的实验成本昂贵的化学实验检测方法更有利于蛋白质复合物相关应用研究的进一步发展。
1.3 国内外研究现状
最早蛋白质复合物的识别方法分为两类:化学实验检测方法、计算方法。
(1)化学实验检测方法。化学实验检测方法已经发展到用实验室里的各种实验来识别蛋白质复合物,例如化学交联,亲和纯化,双杂交等方法。虽然很有潜力,但是基于实验室的方法在效率方面却不能令人满意。就拿亲和纯化法来说,识别不同的蛋白质复合物要求用很多使用不同引诱蛋白的实验。而且对于基于实验室的方法来说,能被它们识别的蛋白质复合物通常是不能完成的,因为有些蛋白质复合物在现有的实验设施下可能不会被发现,而且通过实验获得的蛋白质复合物数目有限而且成本代价较高[[3]]。
(2)计算方法。为了避免化学实验检测方法出现的问题,一些不同的计算方法被提议用来作为识别蛋白质复合物的互补工具。它们大多数都是基于使用各种不同图表的聚类算法。通常来说,为了把PPI网络表示成图表的形式,用图的顶点表示蛋白质,图的边则表示蛋白质之间的相互作用,因此而形成的簇被认为是识别出来的蛋白质复合物。纯粹依赖于蛋白质-蛋白质相互作用网络中的图表的拓扑结构的计算方法通过下列拓扑结构来识别簇:例如密度,k-紧密连接子网和边缘结构。
最近,由于蛋白质的功能信息变得更容易得到,因此提供了一种新的识别蛋白质复合物的方法。对于同样的蛋白质复合物中的蛋白质,它们或许有相似的蛋白质功能信息,所以现在有很多方法都是把蛋白质功能信息考虑在内来识别蛋白质复合物。
- 课题研究内容
1.如何利用html css制作一个美观的上传页面。首先选取一张美观的图作为背景,上传页面主要负责参数文件中各个参数的上传,将这几个参数放在一个大框中,每个参数作为一个小框,通过设置合适的margin属性值使得大框居中,每个小框由参数名和输入框组成,将参数名浮动到做左边并设置为右对齐,然后将输入框浮动到右边,最后添加一个提交按钮并把这个提交按钮的宽度设置为小框的宽度并填充比较美观的背景颜色。
2.如何利用jsp servlet完成数据库的更新、配置参数文件、调用程序运行通知后台给用户发送邮件。首先应该用JDBC连接数据库,然后利用servlet的request对象获取上传页面中输入的各个参数字段,后三个功能是通过bat文件来实现的,配置参数文件要用到echo xxgt;gt;参数文件路径的批处理命令,调用程序运行则要用到java –jar xx.jar 参数文件的批处理命令,结果运行完后会生成一个文件名为clusters的文件,通过检测这个clusters文件是否存在来判断程序有没有运行结束,如果检测到这个文件存在则通知后台给用户发送邮件,后台给用户发送邮件则是通过javamail类,首先应设置发件人邮箱账号和密码(发件人邮箱账号需要开启smtp服务),然后把前台输入的用户邮箱作为收件人邮箱并对其发送邮件。
3.如何利用D3.js将数据可视化。利用D3.JS力学图将数据可视化,将ppi网络中的蛋白质看作结点,将蛋白质之间的相互作用关系看作边,并分别上传到结点数组和边数组中,然后用layout转换数据,最后利用svg中的line画边,circle画顶点。
第2章 DCAFP算法介绍
2.1 算法背景
蛋白质复合物是包含执行不同细胞功能的蛋白质的生物分子。因此,在蛋白质相互作用网络中发现的蛋白质复合物有助于我们理解不同细胞系统中蛋白质的作用,我们对PPI网络进行分析的一个主要目的也正是利用数据挖掘中的聚类分析方法从中找到具有特定功能的蛋白质模块[[4]]。正是因为这个原因,蛋白质复合物识别的问题在过去几十年一直很流行。为了处理它,人们提出了相当多的方法包括化学实验检测方法和计算方法。
化学实验检测方法已经发展到用实验室里的各种实验来识别蛋白质复合物,例如化学交联,亲和纯化,双杂交等方法。虽然很有潜力,但是基于实验室的方法在效率方面却不能令人满意。就拿亲和纯化法来说,识别不同的蛋白质复合物要求用很多使用不同引诱蛋白的实验。而且对于基于实验室的方法来说,能被它们识别的蛋白质复合物通常是不能完成的,因为有些蛋白质复合物在现有的实验设施下可能不会被发现。
为了避免基于实验室方法出现的问题,一些不同的计算方法被提议用来作为识别蛋白质复合物的互补工具。它们大多数都是基于使用各种不同图表的聚类算法。通常来说,为了把PPI网络表示成图表的形式,用图的顶点表示蛋白质,图的边则表示蛋白质之间的相互作用,因此而形成的簇被认为是识别出来的蛋白质复合物。纯粹依赖于蛋白质-蛋白质相互作用网络中的图表的拓扑结构的计算方法通过下列拓扑结构来识别簇:例如密度,k-紧密连接子网和边缘结构。
当然也有一些其他的蛋白质复合物识别聚类算法出现,比如基于蜂群优化机理的信息流聚类模型算法[[5]]和基于模块性的图聚类算法[[6]],然而最近由于蛋白质的功能信息变得更容易得到,因此提供了一种新的识别蛋白质复合物的方法。对于同样的蛋白质复合物中的蛋白质,它们或许有相似的蛋白质功能信息,所以现在有很多方法都是把蛋白质功能信息考虑在内来识别蛋白质复合物。特别地,Lubovac等人引入了两个可供选择的网络措施,结合了功能信息和拓扑属性来加权蛋白质然后根据数值较高的权值来识别蛋白质复合物;Wang等人根据功能信息的相似性评估了蛋白质相互作用网络的可靠性并提出了一个扩展的聚类算法来检测蛋白质复合物;Zhang等人通过合并功能信息在原有的蛋白质相互作用网络上提出了一种增强的网络;Hu等人基于功能信息的独立性给蛋白质相互作用网络加权,然后应用马尔可夫过程来识别蛋白质复合物;Zhang等人提出了一种通用的模型来结合功能信息和拓扑信息;Wu等人利用了多重资源的生物数据,例如基因本体,基因表现图谱和亲和纯化数据两个蛋白质之间的密切关系,并根据密切关系的数值来识别蛋白质复合物。
很明显,前述的方法通过设计不同的措施来使用蛋白质的功能信息从而从不同角度给蛋白质相互作用网络或者蛋白质加权,比如相似性,独立性,概率分布。虽然很有效,但是这些方法都有一个共同的显著的缺点:它们把所有功能信息合并到一起的同时忽略了单个功能信息的参数。对于功能信息,基因本体工程从蛋白质的不同角度把它描述为三个功能种类:生化进程,分子功能,细胞组成。根据我们之前的研究,我们发现对于大多数蛋白质复合物来说它们的蛋白质仅仅在部分功能上相似而不是在所有的功能上都相似。因此,当决定两个蛋白质是否应该被划分到同一个蛋白质复合物的时候,两个蛋白质之间相似的功能种类应该被优先考虑。由于目前的把功能信息考虑在内的方法并不能分辨这些区别,所以我们认为为了识别蛋白质复合物更为准确,充满潜力的蛋白质功能信息有待被挖掘。
在这点上,如果我们有一个方法通过强调相似的功能种类而忽略不相似的功能种类使得识别蛋白质复合物中的蛋白质成为可能,那么识别蛋白质复合物的表现将会大大提升。
为了这样做,我们提出了一个基于下列两个直观的属性可用来识别蛋白质复合物的DCAFP方法[[7]]:
- 相同蛋白质复合物中的蛋白质从拓扑结构的角度来讲是紧密连接的。
- 相同蛋白质复合物中的蛋白质从功能信息的角度来讲至少在部分功能上相似。
可以看出第一个属性要求图聚类应该有一个被广泛应用于识别蛋白质复合物的基于密度的拓扑结构,第二个属性是为了强调考虑单个功能种类参数的必要性。
关于DCAFP方法的实现,我们引入了一个可能性矩阵来代表成对的蛋白质被识别为相同的蛋白质复合物的可能性。如果两个蛋白质之间的可能性值很高就说明它们很可能被识别到相同的蛋白质复合物。使用可能性矩阵的另一个好处是可以识别重叠的蛋白质复合物。为了表示形成蛋白质复合物过程中每个功能种类的参数,我们给每个蛋白质分配了一个相应的参数矢量,利用这个参数矢量,当识别对应的蛋白质属于哪个蛋白质复合物的时候,单个功能种类的参数可以定量地显示。根据前述的两个直观的属性,我们构想出了一个条件最佳化问题并基于可能性矩阵和蛋白质的参数矢量来识别蛋白质复合物。这个最佳化问题被命名为DCAFP,DCAFP采用通过反复的步骤使得可能性矩阵和参数矢量最佳化的策略。这个步骤开始与对所有蛋白质的可能性矩阵和参数矢量的随机猜测,然后反复提高聚类的质量知道集收敛。
DCAFP方法的性能已经通过使用取自酿酒酵母菌和人类两个物种的五个蛋白质相互作用网络和三个基因本体的功能种类被评估。大量实验结果不仅显示了DCAFP方法和目前最先进的方法比较时有极具潜力的表现还说明了DCAFP方法有能力识别重叠的蛋白质复合物,有助于我们系统地分析和全面理解蛋白质之间通过作用完成生命活动的规律[[8]]。
2.2 算法思想及实现
DCAFP算法的思想基于两个直观的属性:(1)相同蛋白质复合物中的各个蛋白质之间从功能角度来讲至少在部分功能上是相似的(2)相同蛋白质复合物中的各个蛋白质从拓扑结构的角度来讲是紧密连接的,因为蛋白质很少作为单一的孤立实体[[9]]。DCAFP算法同时考虑了蛋白质的功能信息和蛋白质相互作用网络中的拓扑结构,把蛋白质复合物识别问题构想成一个条件最佳化问题。
为了实现以上思想,DCAFP算法引入了一个可能性矩阵来代表成对的蛋白质被识别到相同的蛋白质复合物的可能性,如果两个蛋白质之间的可能性值很高就说明它们很可能被识别到相同的蛋白质复合物,使用可能性矩阵的另一个好处是可以识别重叠的蛋白质复合物。为了表示形成蛋白质复合物过程中每个功能种类的参数,我们给每个蛋白质分配了一个相应的参数矢量,利用这个参数矢量,当识别对应的蛋白质属于哪个蛋白质复合物的时候,单个功能种类的参数可以定量地显示。根据前述的两个直观的属性,我们构想出了一个条件最佳化问题并基于可能性矩阵和蛋白质的参数矢量来识别蛋白质复合物:即DCAFP采用通过反复的步骤使得可能性矩阵和参数矢量最佳化的策略。这个步骤开始于对所有蛋白质的可能性矩阵和参数矢量的随机猜测,然后反复提高聚类的质量直到集收敛。具体方法如下:
数学准备工作:
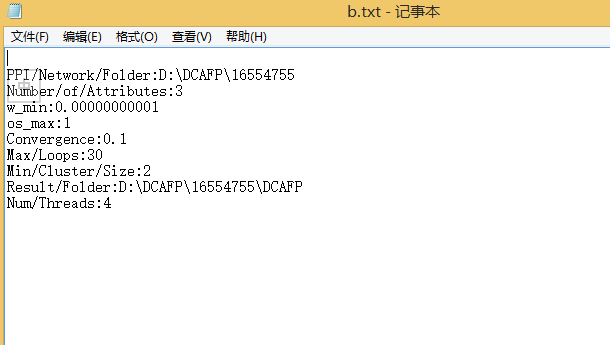
为了表示蛋白质相互作用网络,我们使用了一个三元组G={V,E, Λ},其中V = {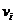 } (
} (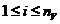 )表示蛋白质结点,E = {
)表示蛋白质结点,E = {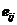 }表示蛋白质结点之间的相互作用关系,Λ = {
}表示蛋白质结点之间的相互作用关系,Λ = {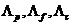 }表示完整的一套三个功能种类,即生化进程,分子功能,细胞组成。任何一个E中的相互作用关系连接G中的两个蛋白质结点。为了表示G的拓扑结构,我们使用了一个邻接矩阵T。对于任意一个功能种类
}表示完整的一套三个功能种类,即生化进程,分子功能,细胞组成。任何一个E中的相互作用关系连接G中的两个蛋白质结点。为了表示G的拓扑结构,我们使用了一个邻接矩阵T。对于任意一个功能种类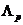 ∈ Λ,我们定义了一个域作为
∈ Λ,我们定义了一个域作为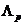 可能会取到的值的集合。在基因本体数据库中,域dom (
可能会取到的值的集合。在基因本体数据库中,域dom (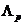 )是
)是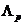 中的一套基因本体条件。
中的一套基因本体条件。
我们使用了一个可能性矩阵W来表示G中所有成对的蛋白质被分组到相同的簇中的可能性大小。通过W的定义,我们使用了一组介于0到1之间的值来表示两个蛋白质结点被识别到相同的簇中的可能性大小,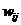 值越高,则蛋白质结点
值越高,则蛋白质结点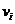 和
和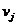 被分组到相同的簇中的可能性就越大。为了表示每个功能种类中蛋白质的相似性,我们使用了一组相似性矩阵A = {
被分组到相同的簇中的可能性就越大。为了表示每个功能种类中蛋白质的相似性,我们使用了一组相似性矩阵A = {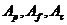 },拿
},拿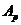 来举例,我们用它来表示生化进程功能的
来举例,我们用它来表示生化进程功能的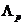 相似性矩阵,
相似性矩阵,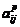 表示就生化进程功能而
表示就生化进程功能而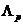 言,两个蛋白质结点之间的相似性大小。
言,两个蛋白质结点之间的相似性大小。
除了W和A矩阵外,我们还有另外一个矩阵D来表示从拓扑结构的角度来讲两个蛋白质之间的相似性。假设对于结点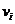 我们有
我们有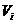 = {
= {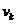 |
|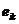 ∈E}来表示跟
∈E}来表示跟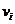 有相互作用关系的结点,同理对于结点
有相互作用关系的结点,同理对于结点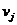 我们也有
我们也有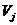 ,
,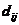 的值表示在
的值表示在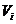 和
和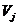 都找到的相同蛋白质的百分比。显然,
都找到的相同蛋白质的百分比。显然,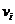 和
和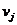 之间相互作用的蛋白质越多,
之间相互作用的蛋白质越多,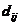 的值就越大。
的值就越大。
以上是毕业论文大纲或资料介绍,该课题完整毕业论文、开题报告、任务书、程序设计、图纸设计等资料请添加微信获取,微信号:bysjorg。
相关图片展示: